De lo que trata esta entrada.
En la entrada no explico a detalles técnicas sobre cada una de los algoritmos empleados, pero pueden consultarse en las referencias o en las categorías Machine Learning en R project y Machine Learning en Python.
Para el ejemplo solo uso código en R, la intención es usar tres muestras de datos simulados para mostrar las estimaciones que realiza la función svm de la librería e1071, solo en el caso de usarla para clasificar. Y por último uso unos datos de correos clasificados como spam o no-spam provenientes del repositorio UCI Machine Learning Repository los cuales se encuentran cargados en la librería kernlab de R project para aplicar las técnicas y comparar svm con otras técnicas.
Ejemplo.-Solo Suppor Vector Machine
En este ejemplo solo hago dos muestras de datos simulados, con distribuciones Gaussianas y la intención es compartir los detalles de qué es en rasgos generales lo que hace el algoritmo de maquina de soporte vectorial al clasificar datos, esto es para dos dimensiones. Es decir, sobre un plano ya que visualmente se vuelve claro y con esa idea se puede pensar en el tipo de cosas que hace el algoritmo en más dimensiones.
Los primeros datos no se mezclan, por lo cual es visualmente claro como separarlos.
#Datos
#Librerías requeridas para el ejemplo
library(ggplot2)
library(e1071)
#Datos de mezcla 1
N<-500
x1<- rnorm(N) * 0.1
y1<- rnorm(N) * 0.1
X1<- t(as.matrix(rbind(x1, y1)))
x2<- rnorm(N) * 0.1 + 0.5
y2<- rnorm(N) * 0.1 + 0.5
X2<- t(as.matrix(rbind(x2, y2)))
X<- as.matrix(rbind(X1, X2))
X=data.frame(X)
X['Clus']=1
X[500:1000,3]=2
ggplot(X,aes(x=x1,y=y1))+geom_point(size=3,aes(colour=factor(Clus)))+ggtitle('Mezcla 1')+theme(plot.title=element_text(lineheight = 2,face='bold'))+
xlab('Variable X1')+ylab('Variable Y1')
La gráfica de los puntos se ve así: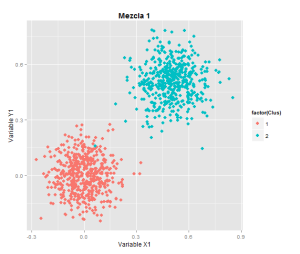
Para la siguiente mezcla considero tres muestras donde las clasifico en dos categorías.
#Mezcla 2
N<-500
x1<- rnorm(500,mean=0,sd=3)*0.1
y1<- rnorm(500,mean=0,sd=3)*0.1
X1<- t(as.matrix(rbind(x1, y1)))
x3<- rnorm(250,mean=0,sd=3)*0.1
y3<- rnorm(250,mean=0,sd=3)*0.1+0.7
X3<- t(as.matrix(rbind(x3, y3)))
x2<- rnorm(N,mean=1.5,sd=4)*0.1 + 0.5
y2<- rnorm(N,mean=0,sd=3)*0.1 + 0.5
X2<- t(as.matrix(rbind(x2, y2)))
Datos2<- as.matrix(rbind(X1, X3,X2))
Datos2=data.frame(Datos2)
Datos2['Clus']=1
Datos2[600:1250,3]=2
ggplot(Datos2,aes(x=x1,y=y1,colour=factor(Clus)))+geom_point(size=3)+ggtitle('Mezcla 2')+theme(plot.title=element_text(lineheight = 2,face='bold'))+
xlab('Variable X1')+ylab('Variable Y1')
 La gráfica de los datos se ve como la anterior imagen.
La gráfica de los datos se ve como la anterior imagen.
Los datos muestran comportamientos distintos, los primeros es claro que se puede separar por una recta, pero la gráfica de la segunda muestra no se ve claramente que lo mejor sea separar los datos por una recta.
Como una buena práctica se debe de tomar una muestra de datos para entrenar el algoritmo (train set) y una cantidad de datos de prueba(test set). Para los siguientes ejemplos considero todos los datos, para ilustrar cual sería la gráfica de los datos predichos por el modelo.
La técnica de SVM para clasificar tienen de fondo la idea de encontrar el mejor «hiperplano» por medio del cual separar los datos. En este ejemplo el concepto de hiperplano es una recta, es decir; los datos están en dos dimensiones (x,y) y se busca la mejor recta que separe los datos, si uno piensa en datos con tres dimensiones (x,y,z) que pueden pensarse como alto, largo y ancho lo que se busca es encontrar el mejor plano que separa los datos, por ello el nombre de hiperplano.
Uso la librería e1071 para estimar el algoritmo SVM con el kernel lineal y radial para la primera muestra de datos, para la segunda uso lineal, polinomial, radial y sigmoid. La intención de este ultimo es compara cual de las cuatro muestra una similitud gráfica más parecida a los datos originales y comparo la tasa de predicciones correctas. Existen más librerías para hacer uso de SVM, dejo en la referencia las ligas.
Primera muestra de datos.
#SVM con Kernel Lineal #Se procede a dejar una semilla para hacer una elección del mejor valor de parámetro cost set.seed (1) #Se hace cross-validation de k-fold, con k=10. Esto mediante la función tune() tune.out=tune(svm,factor(Clus)~.,data=Datos1,kernel="linear",type='C-classification',scale=FALSE,ranges=list(cost=c(0.001,0.01, 0.1, 1,5,10,100))) tune.out summary(tune.out) #Se elige el mejor modelo con el mejor valor para el parámetro cost bestmod=tune.out$best.model summary(bestmod) # Gráfica del mejor modelo con kernel lineal plot(bestmod,Datos1) #Predicción Pred=predict(bestmod,Datos1) table(predicción=Pred,Valores_reales=Datos1$Clus) # Valores_reales # predicción 1 2 # 1 499 1 # 2 0 500 #Con los valores obtenidos se tiene un 99.9% de eficiencia
Algunas observaciones, el modelo pasa por un proceso de validación cruzada usando la función tune(), esto siempre es recomendable para elegir un buen modelo. El parámetro cost, significa el nivel de penalización que permite el modelo, en otras palabras el algoritmo busca la mejor recta que separe los datos y como tal el costo para encontrar esa recta requiere tolerar posibles datos que afectan a la estimación de dicha recta. Esto quizás es una explicación burda y mala, pero detrás de todo algoritmo de Machine Learning está un proceso de optimización el cual determina el valor de los parámetros que hacen que el algoritmo tenga el mejor valor posible.
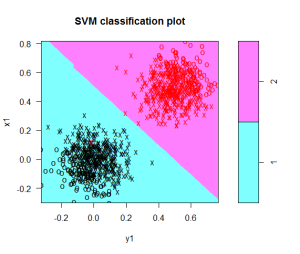
La gráfica que se obtiene de este modelo es la siguiente:
Esta gráfica muestra las dos clases que se buscaban definir.La eficiencia es muy alta, de 1000 datos clasifica correctamente el 99.9%.
Lo que se espera de otro kernel es que prevalezca la eficiencia de SVM y más aún que la gráfica de la clasificación sea casi igual a la obtenido cuando se usa un kernel lineal.
#Kernel radial set.seed (1) tune.out=tune(svm,factor(Clus)~.,data=Datos1,kernel="radial",type='C-classification',scale=FALSE,ranges=list(cost=c(0.001,0.01, 0.1, 1,5,10,100))) tune.out summary(tune.out) bestmod_r=tune.out$best.model summary(bestmod_r) plot(bestmod_r,Datos1) Pred=predict(bestmod_r,Datos1) table(predicción=Pred,Valores_reales=Datos1$Clus) # Valores_reales # predicción 1 2 # 1 499 1 # 2 0 500 #Eficiencia de la clasificación 99.9%
Al obtener la gráfica del modelo por SVM se tiene:
Lo cual muestra una imagen muy similar a la obtenida con el kernel lineal. La tasa de eficiencia al clasificar es prácticamente la misma, 99.9%
Con la segunda muestra de datos pensar en separar por una recta no parece lo natural. Primero hago la estimación con cuatro tipo de kernels de la función svm y muestro la gráfica que regresa el modelo.
#Segunda muestra de datos #Se usa primero el kernel lineal set.seed (1) tune.out=tune(svm,factor(Clus)~.,data=Datos2,kernel="linear",type='C-classification',scale=FALSE,ranges=list(cost=c(0.001,0.01, 0.1, 1,5,10,100))) tune.out summary(tune.out) bestmod_l=tune.out$best.model summary(bestmod_l) plot(bestmod_l,Datos2) Pred=predict(bestmod_l,Datos2) table(predicción=Pred,Valores_reales=Datos2$Clus) # Valores_reales # predicción 1 2 # 1 504 99 # 2 95 552 #Eficiencia de la clasificación 84.4%
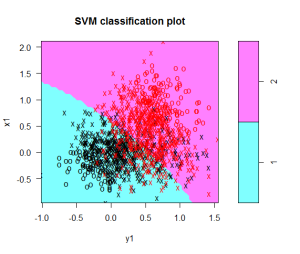
Se tiene la siguiente gráfica como resultado de implementar el algoritmo:
Si se compara los datos que predice el modelo con respecto a los originales se tienen lo siguiente:
Se observa que la predicción muestra totalmente separadas las dos clases y cabe notar que se tiene el 84.4% eficiencia.
#Kernel polinomial set.seed (1) tune.out=tune(svm,factor(Clus)~.,data=Datos2,kernel="polynomial",type='C-classification',scale=FALSE,ranges=list(cost=c(0.001,0.01, 0.1, 1,5,10,100))) tune.out summary(tune.out) bestmod_p=tune.out$best.model summary(bestmod_p) plot(bestmod_p,Datos2) Pred=predict(bestmod_p,Datos2) table(predicción=Pred,Valores_reales=Datos2$Clus) # Valores_reales # predicción 1 2 # 0 543 174 # 1 56 477 #Eficiencia de la clasificación 81.6%, con kernel polinomial
La gráfica que se obtiene del modelo es:
Y otra vez haciendo una gráfica para comparar la predicción con los datos originales se tiene:
Se aprecia que no es tan definida la recta que separa las clases como en el ejemplo del kernel lineal, pero la eficiencia se reduce ya que es de 81.6%
#Kernel sigmoid set.seed (1) tune.out=tune(svm,factor(Clus)~.,data=Datos2,kernel="sigmoid",type='C-classification',scale=FALSE,ranges=list(cost=c(0.001,0.01, 0.1, 1,5,10,100))) tune.out summary(tune.out) bestmod_s=tune.out$best.model summary(bestmod_s) #Informa del tipo de modelo, del que puede considerarse como el mejor modelo plot(bestmod_s,Datos2) Pred=predict(bestmod_s,Datos2) table(predicción=Pred,Valores_reales=Datos2$Clus) # Valores_reales # predicción 1 2 # 1 509 102 # 2 90 549 #Eficiencia de la clasificación 84.6%, con kernel Sigmoid
La gráfica que se obtiene del modelo es la siguiente:
Haciendo la gráfica comparativa de predicción y datos originales se obtiene:
Se aprecia que es muy similar a la que se obtiene con el kernel lineal y hasta el nivel de eficiencia resulta muy aproximado, ya que es del 84.6%
#Kernel Radial set.seed (1) tune.out=tune(svm,factor(Clus)~.,data=Datos2,kernel="radial",type='C-classification',scale=FALSE,ranges=list(cost=c(0.001,0.01, 0.1, 1,5,10,100))) tune.out summary(tune.out) bestmod_r=tune.out$best.model summary(bestmod_r) plot(bestmod_r,Datos2) Pred=predict(bestmod_r,Datos2) table(predicción=Pred,Valores_reales=Datos2$Clus) # Valores_reales # predicción 1 2 # 1 497 94 # 2 102 557 #Eficiencia de la clasificación 84.3%, con kernel lineal
La gráfica obtenida con este kernel es la siguiente:
Y al comparar las predicciones con los datos originales se tiene:
Se aprecia que es similar a la obtenida por el kernel lineal y sigmoid, más aún la eficiencia resulta ser del 84.3%.
Entonces en resumen los datos al ser clasificados mediante SVM con distintos kernel resultó en estos datos resulta tener mejor eficiencia el kernel sigmoid, por un porcentaje mínimo sobre el lineal.
Para mostrar como se comporta SVM con una muestra de datos altamente no lineales o que resulta difícil separar por medio de una recta , genero una muestra más.
#Datos altamente no lineales
#Distribución uniforme 1250 valores
x1=runif(1250)-0.5
x2=runif (1250)-0.5
#Variable Indicadora
y=1*(x1^2-x2^2> 0)
#Gráfica donde se muestran los puntos pintados por etiqueta de y
#Se contruye un data.frame con los datos
X=data.frame(x1,x2,y)
ggplot(data=X,aes(x=x1,y=x2))+geom_point(aes(colour=factor(y),shape=factor(y)))+ggtitle('Datos Originales')+theme(plot.title=element_text(lineheight = 2,face='bold'))+
xlab('Variable X1')+ylab('Variable X2')
La gráfica de los datos es la siguiente:
En esta muestra de datos se aprecia que separar las clases por una recta no parece lo más eficiente, para constatarlo estimo la clasificación con el kernel lineal.
#Kernel lineal set.seed (1) tune.out=tune(svm,factor(y)~.,data=X,kernel="linear",type='C-classification',scale=FALSE,ranges=list(cost=c(0.001,0.01, 0.1, 1,5,10,100))) tune.out summary(tune.out) bestmod=tune.out$best.model summary(bestmod) #Informa del tipo de modelo, del que puede considerarse como el mejor modelo plot(bestmod,X) Pred=predict(bestmod,X) table(predicción=Pred,Valores_reales=X$y) # Valores_reales # predicción 0 1 # 0 305 201 # 1 301 443 #Eficiencia de la clasificación 59.8%
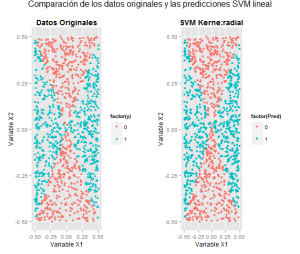
La gráfica que se obtiene del SVM con kernel lineal es:
Haciendo la comparación entre datos originales y las predicciones se tiene lo siguiente:
Se aprecia que no es muy favorable usar el kernel lineal con estos datos, la eficiencia de la clasificación es del 59.4%
Para comparar con otro kernel uso el radial para hacer la clasificación.
#Kernel Radial set.seed(1) tune.out=tune(svm,factor(y)~.,data=X,kernel="radial",sacale=FALSE,type='C-classification',ranges=list(cost=c(0.001,0.01, 0.1, 1,5,10,100))) tune.out summary(tune.out) bestmod=tune.out$best.model summary(bestmod) #Informa del tipo de modelo, del que puede considerarse como el mejor modelo Pred=predict(bestmod,X) table(predicción=Pred,Valores_reales=X$y) # Valores_reales # predicción 0 1 # 0 603 7 # 1 3 637 #Eficiencia de la clasificación 99.2%, con kernel radial
La gráfica que se obtiene con este kernel es la siguiente:
Esta imagen muestra que las curvas que se obtienen con el kernel radial son parecidas a las de los datos originales. Haciendo la comparación entre los datos y las estimaciones se obtiene lo siguiente:
Se aprecia que es muy similar y más aún la eficiencia es del 99.2%, lo cual comparado con el lineal es sumamente mejor usar el kernel radial.
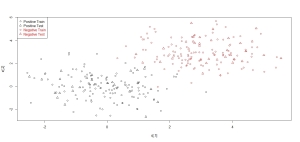
Ejemplo con datos de correos clasificados
Los datos se encuentran cargados en la librería kernelabs, con la cual también se puede estimar la máquina de soporte vectorial. Las comparaciones las hago con varias técnicas:
- Naive Bayes
- LDA y Regresión Logística
- Redes Neuronales
- Árboles de decisión y random forest
Antes de eso aplico con los cuatro kernel disponibles en svm de e1071 para elegir el que mejor clasificación realiza en la muestra de prueba (test set).
Primero reviso los datos.
#Exploración breve de los datos #Se cargan primero las librerías library(kernlab) library(e1071) library(MASS) library(nnet) librery(randomForest) data(spam) #Datos head(spam) dim(spam) #4601 58 str(spam) ############################################# #Preparación de datos index=sort(sample.int(4601,1150)) spam.train=spam[-index,] spam.test=spam[index,]
Se puede hacer algo más al explorar la información, diseñar algunas gráficas o usar alguna técnica de estadística de exploración.
Lo que se hace en el código anterior es dividir los datos entre una conjunto de entrenamiento y un conjunto de prueba.
Aplico los cuatro kernel para elegir el que mejor clasificación realiza.
#SVM con diferentes kernel ######################################## #Kernel lineal svmfit=svm(type~.,data=spam.train,kernel="linear",cost=0.1,type='C-classification' ) summary(svmfit) Pred=predict(svmfit,spam.test) table(predicción=Pred,Valores_reales=spam.test$type) # Valores_reales #predicción nonspam spam # nonspam 646 40 # spam 40 424 #Eficiencia del modelo 93.04% ####################################### #Kernel radial svmfit=svm(type~.,data=spam.train,kernel="radial",cost=0.1,type='C-classification' ) summary(svmfit) set.seed(1) tune.out=tune(svm,type~.,data=spam.train,kernel="radial",type='C-classification',ranges=list(cost=c(0.001,0.01, 0.1, 1,10,100))) tune.out summary(tune.out) bestmod_r=tune.out$best.model summary(bestmod_r) Pred=predict(bestmod_r,spam.test) table(predicción=Pred,Valores_reales=spam.test$type) # Valores_reales #predicción nonspam spam # nonspam 652 36 # spam 34 428 #Eficiencia del modelo 93.91% ################################################# #Kernel sigmoid svmfit=svm(type~.,data=spam.train,kernel="sigmoid",cost=0.1,type='C-classification' ) summary(svmfit) set.seed(1) tune.out=tune(svm,type~.,data=spam.train,kernel="sigmoid",type='C-classification',ranges=list(cost=c(0.001,0.01, 0.1, 1,10,100))) tune.out summary(tune.out) bestmod_s=tune.out$best.model summary(bestmod_s) Pred=predict(bestmod_s,spam.test) table(predicción=Pred,Valores_reales=spam.test$type) # Valores_reales #predicción nonspam spam # nonspam 649 78 # spam 37 386 #Eficiencia del modelo 90% ################################################ #Kernel polynomial svmfit=svm(type~.,data=spam.train,kernel="polynomial",cost=0.1,type='C-classification' ) summary(svmfit) set.seed(1) tune.out=tune(svm,type~.,data=spam.train,kernel="polynomial",type='C-classification',ranges=list(cost=c(0.001,0.01, 0.1, 1,10,100))) tune.out summary(tune.out) bestmod_p=tune.out$best.model summary(bestmod_p) Pred=predict(bestmod_p,spam.test) table(predicción=Pred,Valores_reales=spam.test$type) # Valores_reales #predicción nonspam spam # nonspam 661 53 # spam 25 411 #Eficiencia del modelo 93.2%
Se observa en las tasas de error que el mejor kernel de svm es el radial, así que considero este contra el cual comparo las otras técnicas.
Si bien no explico a detalle lo que hace el código en breve es estimar vía tune() la elección del mejor valor de cross-validation de varios valores.
Una técnica clásica para clasificar correos es Naive Bayes, la cual considera ciertas propiedades teóricas que permiten hacer un calculo rápido. Uso NaiveBayes de la librería e1071.
#Naive Bayes vs SVM elegido NB=naiveBayes(type~.,data=spam.train) Pred=predict(NB,spam.test) table(predicción=Pred,Valores_reales=spam.test$type) # Valores_reales #predicción nonspam spam # nonspam 362 18 # spam 324 446 #Se tiene una eficiencia del 70.2% de bien clasificados
Resulta que es mejor el modelo SVM ante el Naive Bayes por un porcentaje altamente mejor.
#Regresión Logística-LDA vs SVM elegido
Mod.lm=glm(type~.,data=spam.train,family='binomial')
summary(Mod.lm)
Mod.lmprob=predict(Mod.lm,newdata=spam.test,type="response")
Mod.pred=rep('nonspam',1150)
Mod.pred[Mod.lmprob>0.5]='spam'
table(Predicción=Mod.pred,Valores_Reales=spam.test$type)
# Valores_Reales
#Predicción nonspam spam
# nonspam 643 39
# spam 43 425
#Predicción 92.86%
############################################
#Método LDA
lda.fit=lda(type~.,data=spam.train)
lda.pred=predict(lda.fit,spam.test)
lda.class=lda.pred$class
table(Predicción=lda.class,Valores_Reales=spam.test$type)
#lda.class nonspam spam
# nonspam 650 91
# spam 36 373
#Tasa de eficiencia 88.9%
Se tienen que el porcentaje de eficiencia en la clasificación de SVM 93.91% es mayor de casi 1% mejor que los métodos lineales, en este caso la regresión logística resulta ser la mejor entre las técnicas lineales.
Sin explicar detalles elijo un modelo de redes neuronales para clasificar, la librería es nnet y la función tienen el mismo nombre.
#Redes Neuronales red=nnet(type~.,data=spam.train,size=2) Pred=predict(red,spam.test,type="class") table(Predicción=Pred,Valores_Reales=spam.test$type) # Valores_Reales #Predicción nonspam spam # nonspam 650 29 # spam 36 435 #Tasa de eficiencia 94.34%
Resulta que el modelo de redes neuronales resulta ser mejor casi 0.5% mejor que el modelo de SVM. Lo cual faltaría hacer una revisión para definir el mejor modelo de redes neuronales y quizás la mejora es mejor o nula pero tendrías los valores óptimos de los parámetros.
#Dos técnicas de arboles # La primera es por árboles y se hace cross-validation para elegir el mejor modelo tree.spam=tree(type~.,spam.train) set.seed (3) cv =cv.tree(tree.spam,FUN=prune.misclass ) #Gráfica de Cross-validation para elegir el mejor valor de del parámetro best par(mfrow =c(1,2)) plot(cv$size,cv$dev,type="b",col="2") plot(cv$k,cv$dev,type="b",col="3") prune.tree=prune.misclass(tree.spam,best=8) tree.pred=predict(prune.tree,spam.test,type="class") table(tree.pred,Valores_Reales=spam.test$type) # Valores_Reales #tree.pred nonspam spam # nonspam 644 58 # spam 42 406 #Tasa de clasificación 91.3% ################################################# #Modelo de Random Forest set.seed (1) rf.spam =randomForest(type~.,data=spam.train,mtry=7, importance =TRUE) rf.pred_spam=predict(rf.spam,newdata=spam.test,type="class") table(rf.pred_spam,Valores_Reales=spam.test$type) # Valores_Reales #rf.pred_spam nonspam spam # nonspam 663 29 # spam 23 435 #Tasa de clasificación 95.4%
Revisando la tasa de clasificaciones correcta por medio de las técnicas de árboles de decisión , resulta ser la mejor técnica para clasificar los datos de los correos. Se obtienen que la técnica de Random Forest resulta clasificar correctamente el 95.4%, comparado con la red neuronal y SVM es mejor casi en un 2%.
La única intensión de este ejemplo es hacer una comparación entre diversas técnicas de clasificación, de cierto modo hacer un ejemplo sencillo y que se puede replicar. Ha esto le faltaría agregar algunos gráficos que muestran los resultados o hacer algunas variaciones de los parámetros requeridos en cada algoritmos para determinar con mejor eficiencia el mejor modelo.
Espero ilustre de manera breve la variedad de técnicas y permita visualizar que al final se requiere probar con muchos modelos para elegir uno entre todos los realizados.























Debe estar conectado para enviar un comentario.