En la entrada explico la técnica de máquina de soporte vectorial (SVM-Sopport Vector Machine), es un conjunto de algoritmos que pueden aplicarse a problemas de clasificación o regresión.
La explicación no es exhaustiva ni mucho menos menciono aspectos teóricos de la técnica, tan solo trato de mostrar con varios ejemplos como funciona.
Existe mucho material para revisar los detalles y como menciona el autor de la referencia [2], existen ahora nuevas técnicas para clasificar pero es casi obligado en Machine Learning conocer el algoritmo SVM por la relevancia que han tenido.
Una de esas nuevas técnicas es Relevance Vector Machine (RVM), la cual es una variación de SVM en su versión Bayesiana y que permite ver la técnicas SVM desde un punto de vista probabilista.
En otras dos entradas comenté sobre los temas Naive Bayes y Regresión Lineal, la primera técnica es por excelencia una técnica de clasificación y la segunda una de predicción pero se puede desarrollar o modificar para usarla para clasificar.
Las técnicas de SVM cubren ambos aspectos, clasificación y predicción. En la entrada solo comento sobre como usar esta técnica para hacer clasificaciones y muestro un ejemplo sencillo de como usarla para hacer predicción, lo malo de esta familia de técnica es que al construir un modelo uno desea o busca en algunas ocasiones tener una ecuación explicita para analizar el comportamiento de las variables. En este caso eso no es posible, pero para ciertas situaciones no es necesario saber como afectan las variaciones de las variables predictoras, ejemplo para hacer algo de Marketing uno quizás solo busca la clasificación mejor de la muestra o para clasificar textos, quizás no interesa la forma del modelo porque no tenemos una variable respuesta a modelar sino solo entras para clasificar.
Para los ejemplos hago uso de dos librerías en R que calculan los algoritmos de SVM; e1071 y kernlab. También se puede usar para realizar SVM como método de clasificación para dos clases la librería svmpath.
Algunos ejemplos con las librerías
Antes de hacer el ejemplo del texto Machine Learning for Hackers[1] , hago uso de la librería e1071 para mostrar uno de los conceptos claves en SVM, lo que es un hiper-plano. En este caso lo «hiper» se reduce a una recta en el plano. El código es el siguiente:
#Código de muestra de la técnica SVM #Se carga a librería e1071 library(e1071) set.seed(1) #Se generan los datos x=matrix(rnorm(100*2), ncol=2) y=c(rep(-1,10), rep(1,10)) x[y==1,]=x[y==1,] + 1 #Se hace la gráfica de los puntos generados en el plano plot(x, col=(3-y),main="Datos mezclados",xlab="Eje X",ylab="Eje y") #Se construye un Data.Frame dat=data.frame(x=x, y=as.factor(y)) #Se calcula SVM para el kernel lineal svmfit=svm(y~., data=dat, kernel="linear", cost=10) plot(svmfit, dat) #Se puede revisar los valores y el resumen de los estimado en R project para el modelo SVM #svmfit$index #summary(svmfit) #Se estima el modelo SVM con kernel polinomial svmfit=svm(y~., data=dat, kernel="polynomial", cost=10) plot(svmfit, dat) #Se estima el modelo SVM con el kernel sigmoid svmfit=svm(y~., data=dat, kernel="sigmoid", cost=10) plot(svmfit, dat) #Se estima el modelo SVM con el kernel radial svmfit=svm(y~., data=dat, kernel="radial", cost=10) plot(svmfit, dat)
Cuando se corre el código se obtienen cuatro gráficas, la primera solo presenta los datos a clasificar en el plano, la segunda, tercera, cuarta y quinta gráfica muestran la clasificación en dos clases por medio de variaciones al modelo SVM.

Cada gráfica muestra una separación del conjunto de puntos en dos grupos, pese a que no tienen un título cada gráfica que indique el kernel que se usó, la secuencia es correcta con respecto al código. Entonces lo que hace el tipo de kernel es definir un tipo de línea o frontera entre las clases. Se observa que cuando el kernel es lineal se obtienen como frontera entre las clases como la recta de un ajuste lineal. Esa rectas, para el caso lineal la recta es lo que se llama hiperplano.
Para comparar el funcionamiento del algoritmo como técnica de clasificación, hago una comparación con el método Naive Bayes aplicándolo a una muestra 4601 correos electrónico analizados y con una clasificación de ser spam o no. Estos datos se encuentran en la librería kernlab y hago uso de la función naivebayes de la librería e1071.
Los datos son 4601 correos con 58 variables asignadas, es decir; la tabla de datos tienen 4601 renglones y 58 columnas. La última columna indica si el correo fue clasificado como spam o no, esto permite comparar al final la clasificación de una muestra. En este caso se toman 100 correos para evaluar el método SVM kernel radial y polinomial.
#Comparación con otro método de clasificación library(kernlab) data(spam) index=sample(1:nrow(spam),100) fm=ksvm(type~.,data=spam[-index,],kernel="rbfdot",kpar="autolibrary(e1071)matic",C=60,cross=5) pred=predict(fm,spam[index,]) table(pred,spam[index,58]) #Los datos que se obtienen al ejecutar la clasificación son: #pred nonspam spam #nonspam 62 2 #spam 2 34 fm=ksvm(type~.,data=spam[-index,],kernel="polydot",kpar="automatic",C=60,cross=5) pred=predict(fm,spam[index,]) table(pred,spam[index,58]) #Los datos que se obtienen al ejecutar la clasificación son: #pred nonspam spam #nonspam 62 4 #spam 2 32 #Comparación con otro método de clasificación library(e1071) model=naiveBayes(type~.,data=spam[-index,]) pred=predict(mol,spam[index,]) table(pred,spam[index,58]) #pred nonspam spam #nonspam 38 1 #spam 26 35
Calculando el porcentaje de eficiencia de los métodos, SVM tienen 96% y 94% de eficiencia, por otro lado el método Naive Bayes tienen 73% de aciertos. En este ejemplo se observa que el método es eficiente para hacer clasificaciones, en este último ejemplo se tienen 58 variables no todas siguen comportamientos lineales, por lo cual tomar un kernel no lineal para SVM resulta apropiado.
Lo que se observa es que SVM puede servir como un método de clasificación para dos clases, pero también es adecuado para construir un modelo para clasificar para más de dos clases.
El ejemplos del libro Machine Learning for Hackers[1], del cual se pueden descargar los datos en github, inicialmente muestra un comparativo entre un el método de clasificación lineal logit contra SVM.
Hago la regresión logística para los datos que cuentan con una variable indicadora para distinguir entre aceptados y no aceptados y se compara la estimación con el método SVM.
#Cargamos los datos
df <- read.csv(file.path('data', 'df.csv'))
#Calculamos la regresión logística
logit.fit <- glm(Label ~ X + Y,
family = binomial(link = 'logit'),
data = df)
#Estimamos la predicción de los aceptados
logit.predictions <- ifelse(predict(logit.fit) > 0, 1, 0)
#Estimamos la media de la predicción
mean(with(df, logit.predictions == Label))
#[1] 0.5156
El cálculo de SVM se hace mediante la librería e1071.
#Cargamos la librería
library('e1071')
#Estimamos el modelo por medio de SVM
svm.fit <- svm(Label ~ X + Y, data = df)
svm.predictions <- ifelse(predict(svm.fit) > 0, 1, 0)
#Calculamos la media de las predicciones
mean(with(df, svm.predictions == Label))
#[1] 0.7204
Los resultados son los siguientes, el método lineal estima correctamente el 51% de los datos y SVM el 72% .
Para este ejemplo el kernel que se usa para SVM es lineal y sin embargo arroja mejores resultados que la regresión logística.
La gráfica de los resultados se verían así:
#Graficamos los resultados
library("reshape")
df <- cbind(df,
data.frame(Logit = ifelse(predict(logit.fit) > 0, 1, 0),
SVM = ifelse(predict(svm.fit) > 0, 1, 0)))
predictions <- melt(df, id.vars = c('X', 'Y'))
ggplot(predictions, aes(x = X, y = Y, color = factor(value))) +
geom_point() +
facet_grid(variable ~ .)+ggtitle("Ejemplo SVM vs Logit") + theme(plot.title = element_text(lineheight=.9, face="bold"))
En las primeras cuatro gráficas de la entrada mostré que cuando se cambia el tipo de kernel para método SVM las líneas de las clases mediante las cuales separa los datos se comportan de modo distinto. Al aplicar varios tipos de kernel para los datos df, se muestra un gráfico comparativo entre el comportamiento de las fronteras entre los datos.
#Comparamos distintos kernel
#Usamos los datos anteriores
df <- df[, c('X', 'Y', 'Label')]
#Calculamos SVM con el Kernel Lineal
linear.svm.fit <- svm(Label ~ X + Y, data = df, kernel = 'linear')
with(df, mean(Label == ifelse(predict(linear.svm.fit) > 0, 1, 0)))
#Calculamos SVM con el Kernel Polinimial
polynomial.svm.fit <- svm(Label ~ X + Y, data = df, kernel = 'polynomial')
with(df, mean(Label == ifelse(predict(polynomial.svm.fit) > 0, 1, 0)))
#Calculamos SVM con el Kernel Radial o RBF
radial.svm.fit <- svm(Label ~ X + Y, data = df, kernel = 'radial')
with(df, mean(Label == ifelse(predict(radial.svm.fit) > 0, 1, 0)))
#Calculamos SVM con el Kernel Sigmoid
sigmoid.svm.fit <- svm(Label ~ X + Y, data = df, kernel = 'sigmoid')
with(df, mean(Label == ifelse(predict(sigmoid.svm.fit) > 0, 1, 0)))
#Construimos nuestra tabla de predicciones y datos
df <- cbind(df,
data.frame(LinearSVM = ifelse(predict(linear.svm.fit) > 0, 1, 0),
PolynomialSVM = ifelse(predict(polynomial.svm.fit) > 0, 1, 0),
RadialSVM = ifelse(predict(radial.svm.fit) > 0, 1, 0),
SigmoidSVM = ifelse(predict(sigmoid.svm.fit) > 0, 1, 0)))
#Reordenamos nuestros datos
predictions <- melt(df, id.vars = c('X', 'Y'))
#Graficamos los cálculos
ggplot(predictions, aes(x = X, y = Y, color = factor(value))) +
geom_point() +
facet_grid(variable ~ .)
Anteriormente mostré como al evaluar o clasificar correos el método SVM muestra un buen comportamiento al compararlo con el método clásico Naive Bayes para analizar spam/no-spam.
Pero Naive Bayes no es el único método de clasificación entre los algoritmos de Machine Learning, así que hago otro comparativo entre diversas técnicas con datos de 3249 correos detectados como spam y se toman para evaluar los métodos 410 palabras contenidas en ellos. En el ejemplo anterior la base de los correos se encontraba ordenada y procesada, en este caso de hace uso de los textos para detectar y clasificar los correos.
Los datos de los correos se encuentran en formato de una matriz, de la cual se toma el 50% de datos para entrenamiento ( training set) y prueba (test set) el otro 50%.
La cuestión ahora es elegir entre los otros métodos el que resulte mejor como modelo, para eso se hace uso de la validación cruzada y la regularización. Entonces se una el parámetro lambda y el MSE para determinar el lambda apropiado para el modelo logit.
#Cargamos los datos
load(file.path('data', 'dtm.RData'))
set.seed(1)
training.indices <- sort(sample(1:nrow(dtm), round(0.5 * nrow(dtm))))
test.indices <- which(! 1:nrow(dtm) %in% training.indices)
#Definimos los conjuntos de entrenamiento
train.x <- dtm[training.indices, 3:ncol(dtm)]
train.y <- dtm[training.indices, 1]
#Definimos los conjuntos de prueba
test.x <- dtm[test.indices, 3:ncol(dtm)]
test.y <- dtm[test.indices, 1]
#Borramos la matriz original
rm(dtm)
#Cargamos la librería glmnet para calcular logit
library('glmnet')
#Estimamos logit
regularized.logit.fit <- glmnet(train.x, train.y, family = c('binomial'))
lambdas <- regularized.logit.fit$lambda
performance <- data.frame()
#Extraemos los valores de lambda
for (lambda in lambdas)
{
predictions <- predict(regularized.logit.fit, test.x, s = lambda)
predictions <- as.numeric(predictions > 0)
mse <- mean(predictions != test.y)
performance <- rbind(performance, data.frame(Lambda = lambda, MSE = mse))
}
#Graficamos los resultados
ggplot(performance, aes(x = Lambda, y = MSE)) +
geom_point() +
scale_x_log10()
La gráfica que se obtiene es la siguiente:
Ya que se elige el mejor modelo Logit se estima como se comportó al clasificar los correos.
#Resultados de Logit best.lambda <- with(performance, max(Lambda[which(MSE == min(MSE))])) mse <- with(subset(performance, Lambda == best.lambda), MSE) mse #[1] 0.06830769
Se observa que aproximadamente solo el 6% de los correos fueron mal clasificados.
Ahora calculo el modelo SVM para el kernel lineal y radial.
#Calculo de SVM
#Cargamos la librería que se usará
library('e1071')
#Estimamos le modelo para el kernel lineal
linear.svm.fit <- svm(train.x, train.y, kernel = 'linear')
predictions <- predict(linear.svm.fit, test.x)
predictions <- as.numeric(predictions > 0)
mse <- mean(predictions != test.y)
#Calculamos el valor del MSE
mse
#0.128
#Estimamos el modelo para el Kernel radial
radial.svm.fit <- svm(train.x, train.y, kernel = 'radial')
predictions <- predict(radial.svm.fit, test.x)
predictions <- as.numeric(predictions > 0)
#Calculamos MSE
mse <- mean(predictions != test.y)
mse
#[1] 0.1421538
Los dos modelos SVM tienen aproximadamente el 12% y 14% respectivamente de correos mal clasificados. Lo cual es mayor al error de clasificación con el modelo Logit.
Como último ejemplo se hace uso del algoritmo k-vecinos cercanos (k-nn), con k=50.
#Calculamos para 50 vecinos cercanos
#Usamos la librería class para la función knn
library('class')
#Estimamos el modelo
knn.fit <- knn(train.x, test.x, train.y, k = 50)
predictions <- as.numeric(as.character(knn.fit))
#Calculamos MSE
mse <- mean(predictions != test.y)
mse
#[1] 0.1396923
Se observa que este último modelo clasifica incorrectamente aproximadamente el 13 % de los correos.
Para mejorar el uso del modelo k-nn se hace la estimación del modelo para k desde 5 hasta 50 y se elige el mejor modelo, considerando el modelo que tenga el MSE con menor valor.
#Calculamos el k-nn modelo para varios k
performance <- data.frame()
for (k in seq(5, 50, by = 5))
{
knn.fit <- knn(train.x, test.x, train.y, k = k)
predictions <- as.numeric(as.character(knn.fit))
mse <- mean(predictions != test.y)
performance <- rbind(performance, data.frame(K = k, MSE = mse))
}
#Elegimos el mejor k
best.k <- with(performance, K[which(MSE == min(MSE))])
best.k
# 5
#Calculamos MSE para el mejor k
best.mse <- with(subset(performance, K == best.k), MSE)
best.mse
#[1] 0.09169231
Se obtiene que el mejor modelo de la familia k-nn resulta ser para k=5 y aproximadamente hace 9% de malas clasificaciones.
Usando como indicador de la calidad de los modelos al MSE se concluye que el modelo Logit tiene el 6% de errores. Así que el comportamiento de esta muestra de correos parece ser mejor modelado por técnicas lineales que no-lineales.
Lo que se observa con el ejemplo es que uno debe de realizar varios modelos para el mismo conjunto de datos y debe de tener algún criterio o indicador para determinar cual es el mejor modelo de entre todos los realizados. No debe uno de creer que una familia de modelos es mejor que la otra, solo alguna funciona mejor para ciertos datos que otra y nada más.
Siempre es conveniente tener más de un modelo y en caso de que dos puedan ser adecuados, es valido hacer ligeras variaciones de esos para determinar cual se adecua mejor al tipo de datos o fenómeno analizado.
Como ejemplo del tipo de gráficas que se obtiene de la librería kernlab hago un ejemplo. Cabe mencionar que esta librería y e1071 cuentan con varias funciones que concentrar algoritmos usuales en Machine Learning. Lo que las distingue es que la primera, kernlab; hace un concentrado los algoritmos dependientes de algún tipo de kernel.
Para el ejemplo se crean 300 puntos y se clasifican en dos grupos, análogo a los primeros ejemplos se busca la curva que separa en dos clases a los datos.
#Generamos los datos
n<-300 #Número de puntos
p<-2 #Dimensiones
sigma<-1 #Varianza de la distribución
meanpos<-0 #Centro de la distribución de los puntos positivos
meanneg<-3 #Centro de la distribución de los putnos negativos
npos<-round(n/2) #Número del ejemplo positivos
nneg<-n-npos #Números negativos del ejemplo
#Generación de los postivos y negativos
xpos<-matrix(rnorm(npos*p, mean=meanpos,sd=sigma),npos,p)
xneg<-matrix(rnorm(nneg*p, mean=meanneg,sd=sigma),nneg,p)
x<-rbind(xpos,xneg)
#Generación de etiquetas
y<-matrix(c(rep(1,npos),rep(-1,nneg)))
#Visualización de datos
plot(x,col=ifelse(y>0,1,2))
legend("topleft",c('Positivos','Negativos'),col=seq(2),pch=1,text.col=seq(2))
 La gráfica muestra la mezcla de puntos en color rojo y negro. Primero se estima el modelo lineal de SVM para determinar la clasificación, en este caso se toma un subconjunto de entrenamiento y otro de prueba.
La gráfica muestra la mezcla de puntos en color rojo y negro. Primero se estima el modelo lineal de SVM para determinar la clasificación, en este caso se toma un subconjunto de entrenamiento y otro de prueba.
#Separamos los datos en entrenamiento y prueba
ntrain <- round(n*0.8) #Entrenamiento
tindex <- sample(n,ntrain) #Muestra de índices para entrenamiento
xtrain <- x[tindex,]
xtest <- x[-tindex,]
ytrain <- y[tindex]
ytest <- y[-tindex]
istrain=rep(0,n)
istrain[tindex]=1
#Visualización
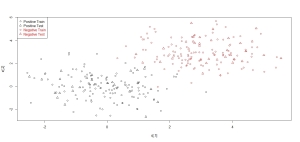
plot(x,col=ifelse(y>0,1,2),pch=ifelse(istrain==1,1,2))
legend("topleft",c('Positive Train','Positive Test','Negative Train','Negative Test'),col=c(1,1,2,2),pch=c(1,2,1,2),text.col=c(1,1,2,2))
library(kernlab)
#Entrenamiento de SVM
svp <- ksvm(xtrain,ytrain,type="C-svc",kernel='vanilladot',C=100,scaled=c())
#Graficación
plot(svp,data=xtrain)
En la primera gráfica se muestra como se comportan los subconjunto de entrenamiento y prueba. En la segunda gráfica se muestra como el método SVM separa los datos y se queda con los negritos dentro de lo que se conoce como margen. Las clases en las cuales separa los datos les da color rojo y azul. En general cada librería cuenta con algunos datos los cuales se encuentran procesados y son un recurso para probar con las funciones y métodos.
Un ejemplo para usar SVM para regresión
Para este ejemplo uso datos de R Project correspondientes a un indicador financiero Alemán; DAX, estos corresponden a los cierres entre 1991 y 1998.
#Se cargan los datos
data("EuStockMarkets")
#Exploramos los datos
head(EuStockMarkets)
#Se toma solo solo el indicador DAX y se construye
#una variable para el tiempo, t
t=1:length(EuStockMarkets[,1])
M=data.frame(EuStockMarkets[,1],t)
colnames(M)<-c("DAX","Tiempo")
#Se construye el modelo con SVM
Modelo=ksvm(M$Tiempo,M$DAX)
#Se hace la gráfica del modelo
plot(M$Tiempo,M$DAX,type="l", main="Ejemplo de SVM para regresión", xlab="Tiempo",ylab="DAX")
lines(M$Tiempo,predict(Modelo,M$Tiempo),col="red")
 La imagen muestra el resultado que predice un modelo regresivo con la familia de técnicas de SVM, este modelo no es el mejor, ya que se tendría que explorar el comportamiento del indicador DAX y hacer algunas pruebas para elegir el mejor kernel para este modelo. Pero la intención es mostrar que SVM también puede ser usado para construir modelos regresivos, la desventaja ante modelos de regresión es la expresión o ecuación, pero como mencioné anteriormente esto realmente es relevante dependiendo de los datos y el origen de la necesidad de modelar estos datos.
La imagen muestra el resultado que predice un modelo regresivo con la familia de técnicas de SVM, este modelo no es el mejor, ya que se tendría que explorar el comportamiento del indicador DAX y hacer algunas pruebas para elegir el mejor kernel para este modelo. Pero la intención es mostrar que SVM también puede ser usado para construir modelos regresivos, la desventaja ante modelos de regresión es la expresión o ecuación, pero como mencioné anteriormente esto realmente es relevante dependiendo de los datos y el origen de la necesidad de modelar estos datos.
Espero sirva de guía lo mostrado en la entrada, en las referencias se encuentran muchos detalles que no menciono.
Referencias:
1.-http://shop.oreilly.com/product/0636920018483.do
2.-http://www.amazon.com/Machine-Learning-Probabilistic-Perspective-Computation/dp/0262018020
3.-http://research.microsoft.com/en-us/um/people/cburges/papers/svmtutorial.pdf
4.-http://www.springer.com/gp/book/9780387987804
5.-http://www.kernel-machines.org/
6.-http://www.support-vector-machines.org/
7.-http://www.csie.ntu.edu.tw/~cjlin/libsvm/
8.-El capítulo 7, http://www.springer.com/gp/book/9780387310732
9.-El capitulo 12, http://statweb.stanford.edu/~tibs/ElemStatLearn/









hola es un excelente post, no se si me puedas pasar el contenido del archivo «datadf.csv», para seguir con mayor atención tu post, estoy viendo la clasificación de textos (muy cortos) con SVM en R y esto me ayudaría a empezar a abordar mi problema ya que si ando medio perdido. de antemano gracias y saludos!!.
Te envié el archivo a tu correo, en caso de que tengas duda de como se genera ese archivo puedes consultar en la entrada sobre la clasificación Naive Bayes y el procesamiento de textos. Saludos!
muchas gracias 🙂 disculpa las molestias.
hola de nueva cuneta, en el ejemplo del SPAM me sale este error cuando ejecuto el siguiente comando: > logit.fit <- glm(Label ~ X + Y, family = binomial(link = ‘logit’), data = df)
Error in eval(expr, envir, enclos) : object ‘Label’ not found
no se si me puedas ilustrar para sacar este error.
Rafael, carga los datos df.csv desde el directorio donde los estas guardando, el error es debido a que glm() no está identificando la columna ‘Label’ en los datos df en R. Para verificar que esté el data.frame correctamente cargado para esto desplaza en R hasta el directorio donde están los datos y usa read.csv( ‘df.csv’). Para ver que está bien cargado puedes revisar esto con head(df) y te debe de mostrar 3 columnas, la tercera con nombre ‘Label’.
This looks amazing! I wish you wrote in English. I can understand the code but would have loved to go through the explanation as well. Please get back to me on my email id. Its on my blog. Thanks!
Cordial saludo,
Excelente, muy bien explicado, muchas gracias, Queria pedirte el favor si me puedes enviar los datos para el ejercicio, “datadf.csv
Si, con gusto. Sería más fácil si me escribes al correo d.legorreta.anguiano@gmail.com
Saludos!